Acidi nucleici
Vi è una certa analogia strutturale fra acidi nucleici e proteine. Cosi come le
proteine sono polimeri lineari di amminoacidi, gli acidi nucleici sono polimeri lineari i
cui mattoni costituenti sono i nucleotidi. Queste unità monomere possono
essere ottenute per degradazione in condizioni blande degli acidi nucleici, mentre
un'idrolisi completa (acida, basica o enzimatica) ne fornisce i componenti fondamentali:
questi sono rappresentati da uno zucchero (il D-ribosio nel caso dell'RNA e il
deossiribosio nel caso del DNA, entrambi in forma furanosica), una base eterociclica
(purinica o pirimidinica) e uno ione fosfato. La struttura del
![]() -D-ribosio e del
-D-ribosio e del
![]() -2-deossi-ribosio è mostrata in Figura 20
(il termine deossi sta semplicemente a indicare che il gruppo OH in posizione 2 è
sostituito da un idrogeno).
-2-deossi-ribosio è mostrata in Figura 20
(il termine deossi sta semplicemente a indicare che il gruppo OH in posizione 2 è
sostituito da un idrogeno).
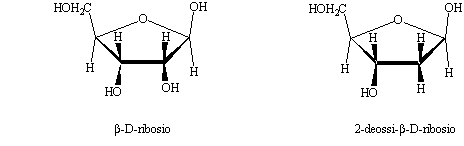
Figura 20. Struttura del ![]() -D-ribosio (RNA)
e del 2-deossi-
-D-ribosio (RNA)
e del 2-deossi-![]() -D-ribosio (DNA).
-D-ribosio (DNA).
Le quattro basi che si ottengono per idrolisi del DNA sono invece mostrate in Figura 21.
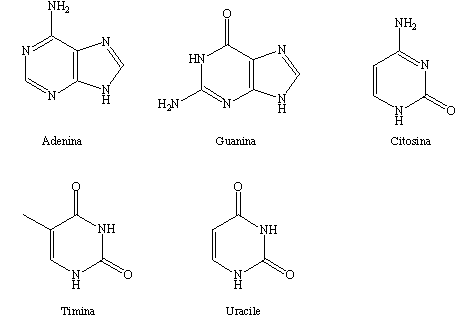
Figura 21. Le basi presenti nel DNA e RNA.
Esse sono l'adenina, la guanina, la citosina
e la timina. Le basi che si ottengono dall'RNA sono le stesse eccettuata la
timina che è sostituita dall'uracile, anch'esso mostrato in Figura 21.
Un nucleoside si ottiene combinando una molecola di D-ribosio (RNA) o di
2-deossiribosio (DNA) con una base eterociclica azotata tramite un legame
![]() -N-glicosidico.
-N-glicosidico.
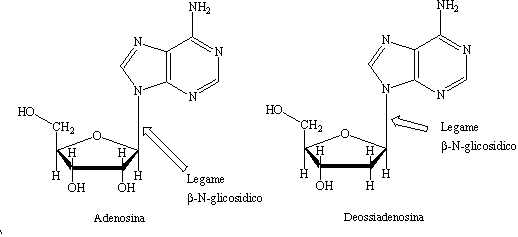
Figura 22. Struttura di due nucleosidi: adenosina (RNA) e deossiadenosina (DNA).
In Figura 22 sono mostrate le strutture di due nucleosidi: uno derivato dall'RNA ed uno dal DNA. Il primo (adenosina) contiene infatti come unità mosaccaridica il D-ribosio legato alla base adenina e il secondo (deossiadenosina) contiene il 2-deossi-D-ribosio legato alla medesima base. Gli altri nucleosidi associati al DNA sono la deossiguanosina (2-deossi-D-ribosio + guanina), la deossicitidina (2-deossi-D-ribosio + citosina), la deossitimidina (2-deossi-D-ribosio + timina). Analogamente, i rimanenti nucleosidi per l'RNA sono la guanosina (D-ribosio + guanina), la citidina (D-ribosio + timina) e l'uridina (D-ribosio + uracile). Un nucleotide, che costituisce la vera unità monomera dell'acido nucleico, può essere ottenuto da un nucleoside esterificando una molecola di acido fosforico con un ossidrile libero della componente monosaccaridica, normalmente l'ossidrile attaccato al carbonio C3' o C5' (nei nucleotidi gli atomi di carbonio dello zucchero sono indicati con i numeri 1', 2', 3', ...). La struttura generale di un nucleotide del DNA, in cui l'esterificazione è avvenuta sul gruppo OH in posizione 5', è mostrata in Figura 23.
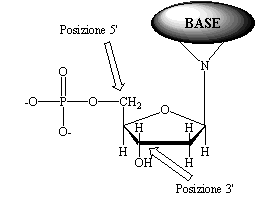
Figura 23. Struttura generale di un nucleotide. Sono messe in evidenza le posizioni
3' e 5' sulle quali puo' avvenire l'esterificazione.
Nel seguito di questa sezione focalizzeremo la nostra attenzione soprattutto sul DNA di cui esamineremo a grandi linee la struttura fondamentale e il meccanismo di replicazione.
1. La struttura primaria degli acidi nucleici
Esaminiamo ora la sequenza con cui i vari monomeri si succedono lungo la catena di un acido nucleico a formare lo scheletro covalente o struttura primaria del polimero. Se consideriamo un filamento di DNA, lo scheletro è costituito da unità alternate di deossiribosio legato ad una base (nucleoside), e di fosfato. L'acido fosforico è doppiamente esterificato e forma un ponte fra il carbonio C3' di un nucleoside e il carbonio C5' di un altro nucleoside. Le basi eterocicliche, legate al carbonio anomerico C1', si distaccano dall'ossatura del polimero costituita dall'alternarsi di unità di acido fosforico e di zucchero. Una rappresentazione schematica della struttura primaria di un tratto di filamento di DNA è riportata in Figura 24. La sequenza delle basi, ovvero l'ordine con cui le varie basi si succedono lungo la catena, può essere specificato elencando semplicemente le lettere che indicano le singole basi, partendo, a sinistra, dall'estremità della catena con il gruppo OH in posizione 5' libero e procedendo verso destra in direzione della posizione 3' libera. I simboli che vengono utilizzati per le varie basi sono le lettere (maiuscole) iniziali del nome: A=adenina, G=guanina, C=citosina, T=timina, U=uracile.
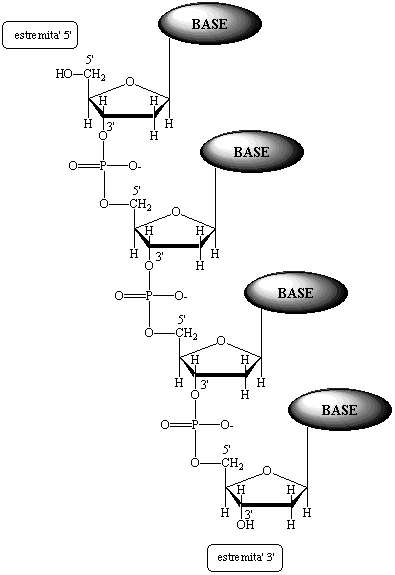
Figura 24. Struttura primaria del DNA. La base puo' essere citosina, timina, adenina,
guanina.
2. Struttura secondaria del DNA
Come nel caso delle proteine anche gli acidi nucleici sono caratterizzati da una struttura secondaria. Un modello per la struttura secondaria del DNA fu proposto nel 1953 da J.D. Watson e F.H.C. Crick e fu confermato nel 1962 da M. Wilkins tramite tecniche ai raggi X. Secondo questo modello due lunghe catene di DNA sono avvolte in una doppia elica attorno allo stesso asse. Le due catene polimeriche sono disposte parallelamente l'una all'altra, ma corrono in direzione opposta: questo significa che a entrambe le estremità della doppia elica è presente l'estremità 5' di un filamento e l'estremità 3' dell'altro filamento. Le due eliche destrorse sono orientate in modo tale da avere lo scheletro di zucchero e fosfato nella parte esterna della struttura con le basi rivolte verso l'interno. Legami idrogeno fra le basi tengono insieme la doppia elica. L'ampiezza massima della spirale è di 20 Å. Dieci coppie successive di nucleotidi, con dieci coppie di basi, danno luogo ad un giro completo della spirale. Due diverse rappresentazioni molto schematiche dei due filamenti della doppia elica che corrono paralleli, ma in senso opposto, con le varie basi accoppiate fra di loro, sono date in Figura 25.
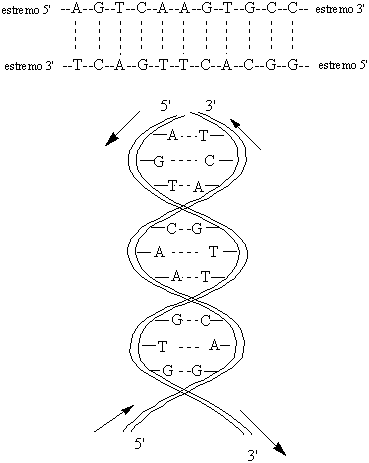
Figura 25. Due rappresentazioni schematiche del modello a doppia elica del DNA.
Sono evidenti gli accoppiamenti fra le varie basi.
I legami idrogeno che tengono insieme la doppia elica non possono formarsi in modo casuale, ma devono formarsi solo fra specifiche coppie di basi. Le coppie consentite nel caso del DNA sono: adenina-timina (A-T) e citosina-guanina (C-G). Le due basi adenina e timina possono interagire tramite due forti legami idrogeno, mentre citosina e guanina possono formare tre legami idrogeno. L'effetto stabilizzante complessivo dovuto a queste interazioni è di circa 10 kcal/mol nel primo caso e circa 17 kcal/mol nel secondo. Le due coppie di basi considerate con l'indicazione dei legami idrogeno e i valori dei relativi parametri strutturali sono mostrate in Figura 26. Altri accoppiamenti non sono consentiti perché, o si avrebbero coppie di basi troppo voluminose o i legami idrogeno che si verrebbero a formare non sarebbero altrettanto forti e stabilizzanti. Questo fatto fa sì che le due catene della doppia elica siano del tutto complementari: ogni volta che una molecola di adenina (A) compare in una catena, una timina (T) deve essere presente nella corrispondente posizione sulla catena opposta. In modo analogo una guanina (G) da una parte implica la presenza di una citosina (C) dall'altra. Questo carattere complementare delle catene è evidente dai due schemi di Figura 25.
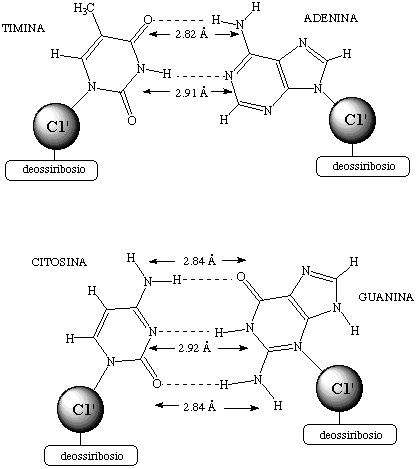
Figura 26. Legami idrogeno per le due coppie di basi adenina-timina e citosina-guanina.
È indicato per ogni base il legame con il C1' dell'unita' di deossiribosio.
3. La replicazione del DNA
Il DNA immagazzina, come già detto, il codice genetico nelle cellule viventi. L'informazione genetica è codificata nelle sequenze particolari di basi presenti che determinano, a loro volta, l'esatta sequenza in cui i vari amminoacidi devono comparire nelle proteine sintetizzate come, ad esempio, un enzima. La trasmissione del codice genetico da una cellula all'altra avviene attraverso una duplicazione esatta della catena del DNA. La replicazione del DNA implica la separazione dei due filamenti della doppia elica, ognuno dei quali funziona come un vero e proprio stampo che determina la sequenza di basi dei due nuovi filamenti che si vanno formando. Immediatamente prima della divisione cellulare, la doppia elica di DNA comincia a svolgersi ad una delle due estremità e su ciascuna delle due catene iniziano a formarsi le due catene complementari. Questo tipo di replicazione, schematizzata in Figura 27, è considerata di tipo conservativo poiché in ognuna delle due nuove doppie eliche è presente un filamento proveniente dall'elica originale. In realtà, la replicazione implica una serie piuttosto complessa di reazioni catalizzate da enzimi diversi. A titolo di esempio, l'enzima DNA-polimerasi catalizza la reazione di addizione di nuovi nucleotidi alla catena in crescita.
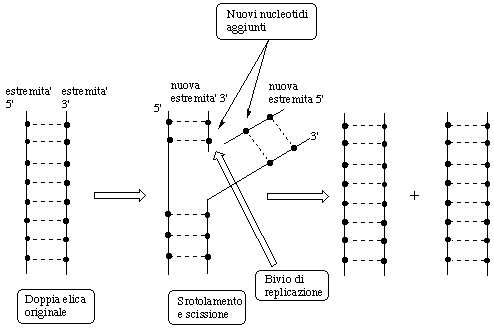
Figura 27. Il processo di replicazione del DNA.
La sintesi di entrambe le due nuove catene di DNA avviene nella direzione
(estremità 5') ![]() (estremità 3').
Ricordando che le due catene
originali corrono parallele, ma in direzioni opposte, si ha come conseguenza che al
bivio di replicazione (vedi Figura 27) una delle due nuove catene deve presentare libero
un OH all'estremità 3' e l'altra nuova catena un OH all'estremità 5'.
È interessante notare che, mentre il complementare del
filamento 3'
(estremità 3').
Ricordando che le due catene
originali corrono parallele, ma in direzioni opposte, si ha come conseguenza che al
bivio di replicazione (vedi Figura 27) una delle due nuove catene deve presentare libero
un OH all'estremità 3' e l'altra nuova catena un OH all'estremità 5'.
È interessante notare che, mentre il complementare del
filamento 3' ![]() 5' viene
sintetizzato come una catena continua senza interruzioni, il complementare dell'altro
filamento viene prodotto come una serie di corte catene. Enzimi specifici provvedono poi
a saldare fra di loro questi segmenti e a formare il nuovo filamento completo di DNA.
5' viene
sintetizzato come una catena continua senza interruzioni, il complementare dell'altro
filamento viene prodotto come una serie di corte catene. Enzimi specifici provvedono poi
a saldare fra di loro questi segmenti e a formare il nuovo filamento completo di DNA.